Transcriptomul SARS-CoV-2: prima hartă a organizării ARN-ului genomic și subgenomic. Cum sunt exprimate proteinele virale și care sunt posibilele aplicații pentru prevenție și tratament?
Genomul noului coronavirus a fost descifrat la doar o lună de la apariția primelor cazuri în Wuhan. Deși genomul a fost secvențiat rapid, au rămas întrebări legate de poziția exactă și rolul anumitor gene. Cele 29 de proteine pe care noul coronavirus le produce au fost deja caracterizate în detaliu în literatură, la fel și genomul lung de aproape 30.000 de nucleotide. Dar aceste date nu sunt suficiente pentru a înțelege complexitatea componentelor și mecanismelor virale. Transcriptomica mai aduce un nivel de informație și permite identificarea de noi ținte.
Atunci când SARS-CoV-2 intră într-o celulă, materialul său genetic – o moleculă lungă de ARN – este replicat (ARN-ul genomic). În același timp se generează și un set de molecule mai mici care alcătuiesc ARN-ul subgenomic.
O hartă a structurii SARS-CoV-2 la rezoluție înaltă a fost publicată recent în Cell, de o echipă din Seoul, din cadrul Center for RNA Research, Institute for Basic Science (IBS) în colaborare cu Korea National Institute of Health (KNIH). Această hartă cuprinde o analiză a transcriptelor (molecule de ARN) produse de SARS-CoV-2 în celulele gazdă, folosind tehnici avansate de secvențiere – secvențiere directă ARN de tip nanopore și secvențiere ADN nanoball. Acestea sunt metode complementare care permit caracterizarea genomului noului coronavirus cu acuratețe.
Rezultatele cercetării sunt extrem de importante pentru că ar putea genera noi direcții pentru a înțelege mecanismele noului coronavirus și implicit ar facilita dezvoltarea de tratamente sau metode de prevenție.
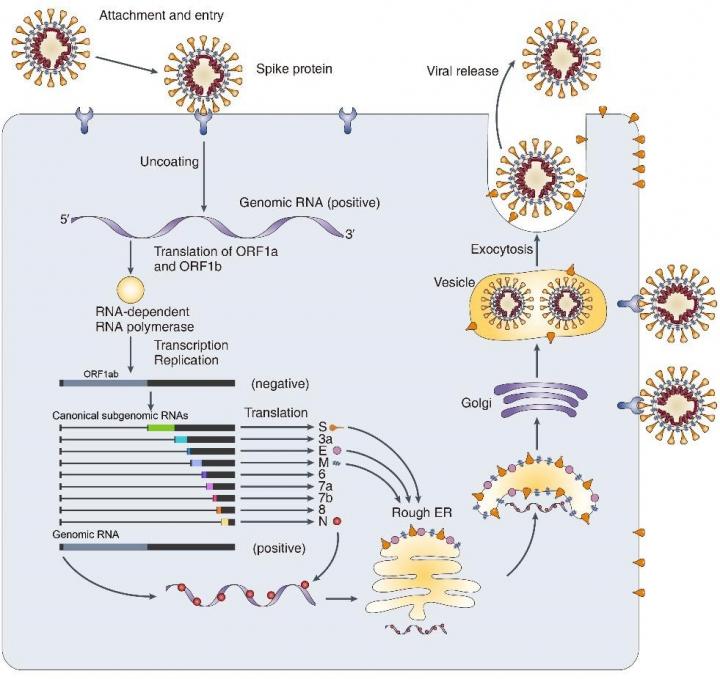
Studiile realizate până acum, în care s-a analizat secvența ARN, au putut doar să facă o estimare asupra localizării genelor. Dincolo de această descriere, analiza setului complet de ARN și înțelegerea organizării genomului SARS-COV-2 este esențială pentru dezvoltarea de strategii pentru diagnostic și tratament. Noul studiu a analizat cum se exprimă informația de la nivelul fiecărui ARN și a oferit noi date despre localizarea genelor.
Harta organizării genomice și subgenomice a noului coronavirus cuprinde câteva informații cheie:
- Analiza detaliată a întregului transcriptom (seturile de ARN ) sugerează un nivel de complexitate ridicat;
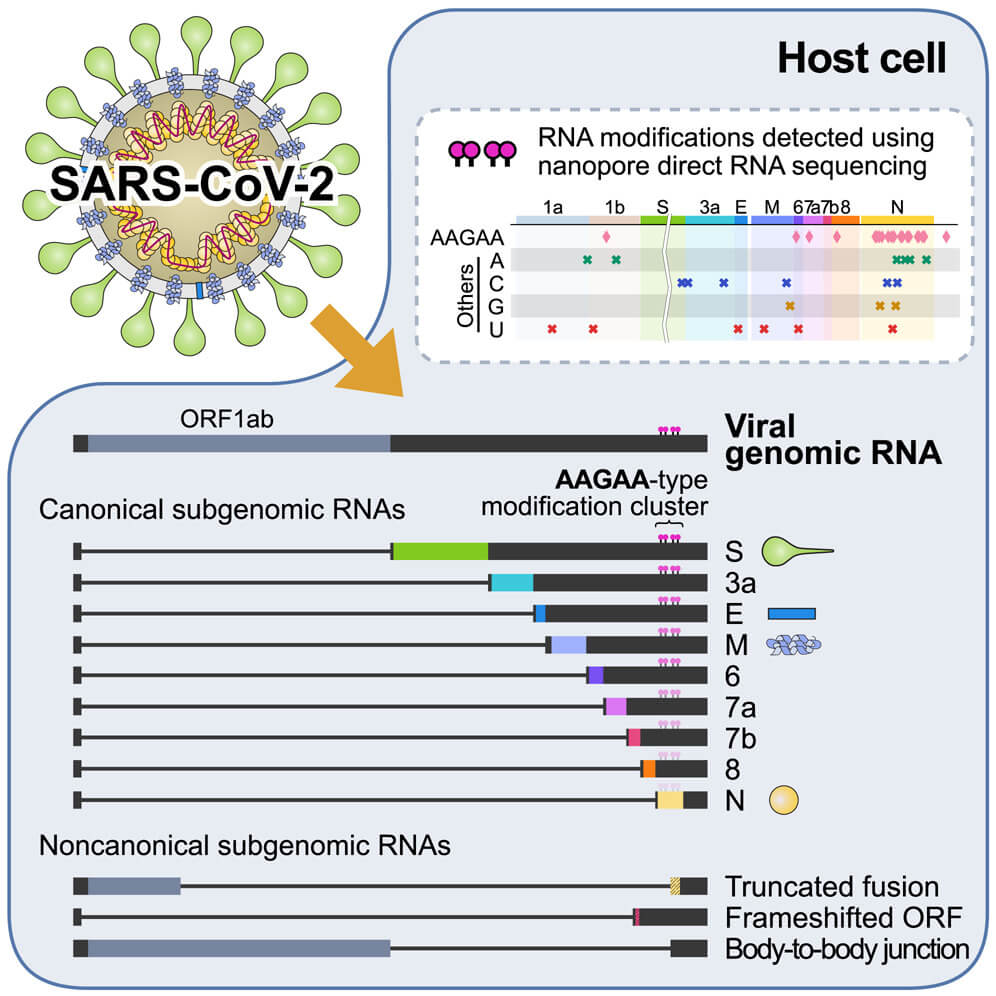
- Particulele virale conțin doar 9 tipuri de ARN subgenomic (mesajele care codifică proteinele structurale, care alcătuiesc „învelișul” viruslui), nu 10, cum se estima anterior (ORF 10 nu e exprimat);
- Analiza epitranscriptomului (modificările biochimice de la nivelul lanțurilor de ARN care nu modifică secvența nucleotidelor) oferă noi argumente pentru evoluția rapidă a virusului și sugerează mecanisme prin care virusul rezistă la atacul imun al gazdei.
Cum e organizat noul coronavirus?
Genomul unui organism reprezintă un set complet de instrucțiuni care dictează proteinele care vor fi produse, dar și modul în care acestea vor fi exprimate. Prin transcriere se face o copie ARN a unei secvențe genomice. Copia se numește ARN mesager. Acesta părăsește nucleul și intră în citoplasmă unde direcționează sinteza proteinelor. Translația este procesul prin care mesajul de la nivelul moleculei de ARN e transformat în secvența de aminoacizi care alcătuiesc proteinele.
Virusurile sunt doar „vești proaste înfășurate în proteine (pieces of bad news wrapped in proteins)”, comenta unul dintre laureații Premiului Nobel pentru Medicină din 1960, Peter Medawar.
SARS-CoV-2 are genomul organizat sub forma unei molecule foarte lungi de acid ribonucleic – ARN. Când infectează o celulă și se replică, apar și alte moleculele mai mici de ARN. Identificarea locului fiecărei gene de la nivelul tuturor tipurilor de ARN virale posibile permite descifrarea acestei „vești proaste”, iar harta propusă de cercetătorii japonezi poate ghida o strategie de combatere a noului coronavirus.
SARS-CoV-2 face parte din genul betacoronavirus împreună cu SARS-CoV și MERS-CoV. Coronavirusurile au cele mai mari genomuri dintre toate virusurile din familia ARN. Cea mai mare parte din genom (două treimi) codifică gene pentru polimeraza virală și alte proteine nestructurale (ORF1a, ORF1b). O treime din genom corespunde unor proteine structurale – proteina spike (S), proteine care fac parte din anvelopa virală (E), membrana (M), nucleocapsida (N) și alte proteine helper. Acestea sunt codificate de ARN subgenomic. ARN-ul genomic e împachetat cu ajutorul proteinelor structurale pentru a forma noii virioni.
Ce înseamnă ORF?
Codul genetic este „citit” în grupuri de 3 nucleotide (unitățile de bază, din care sunt formați acizii nucleici – ADN-ul și ARN-ul). Un astfel de grup se numește codon. La nivelul ribozomilor, unde sunt sintetizate proteinele, acești codoni sunt interpretați, iar fiecărui codon îi corespunde un aminoacid. ORF descrie o secvență neîntreruptă de codoni – de la codonul start la codonul stop.
Open reading frame (cadrul de citire) este termenul folosit pentru o secvență de nucleotide care, atunci când e decodificată și transformată în secvența de aminoacizi, nu conține codoni stop. Prin termenul „open” (deschis) se descrie faptul că ARN-ul poate fi citit în continuare, adăugând aminoacid după aminoacid.
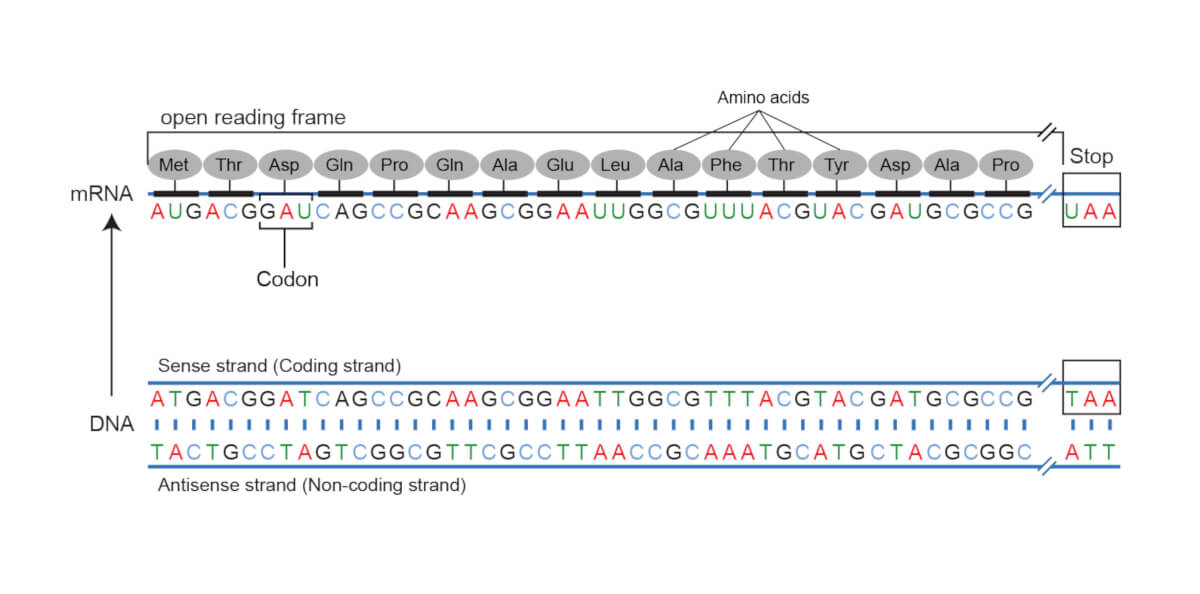
Dar regulile după care se citește codul genetic sunt mult mai complicate. Și sensul în care e citit e important. O moleculă de ADN are două lanțuri complementare, aflate în oglindă. Ambele lanțuri sunt citite în direcția 5` – 3`. Pentru un lanț, există 3 cadre posibile de citire. În total, pentru orice regiune ADN, sunt posibile 6 cadre: 3 în direcția de citire și 3 în sens opus. Odată ce o genă a fost secvențiată este important să se determine cadrul de citire corect.
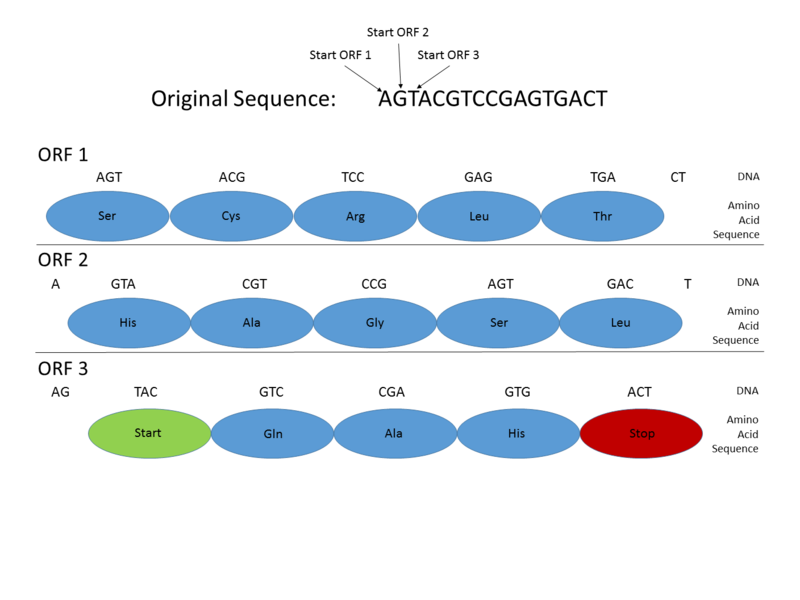
în ARN și apoi interpretată la nivelul ribozomilor in aminoacizi Sursa foto – Wikiwand
În biologia moleculară, aceste concepte au un rol important în identificarea genelor. SARS-CoV 2 produce un număr mare de transcripte care codifică ORF cu rol necunoscut, care prezintă fenomene multiple de fuziuni, deleții, schimbări de cadru.
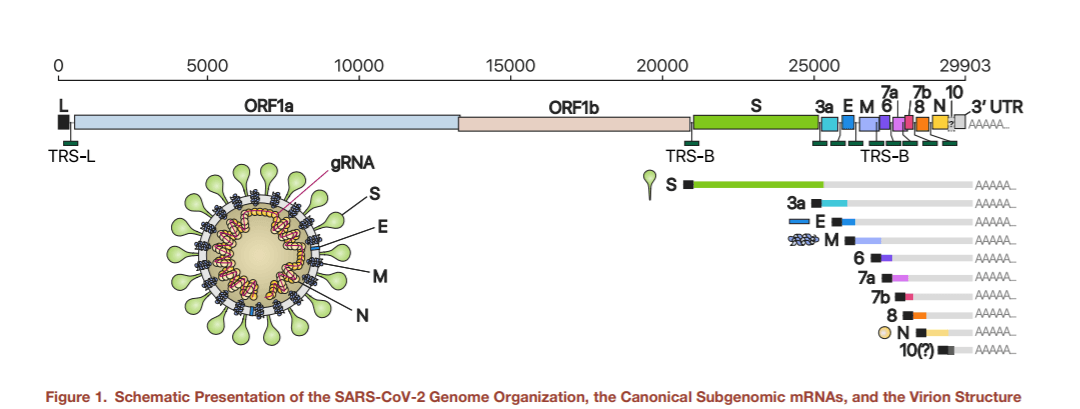
Întregul genom al virusului – ARN-ul genomic are aproximativ 30.000 de nucleotide (prin comparație, genomul uman are 3 miliarde). Atunci când găsește o celulă pe care o poate infecta, virusul introduce o molecula lungă de ARN, întregul genom. Informația e folosită pentru a produce proteine nestructurale. Primele două cadre de citire (ORF1a/b), reprezentând două treimi din lungimea genomului codifică 16 proteine nestructurale. Celelalte ORF sunt la capătul 3 si codifică proteine structurale.
Aceasta conduce la formarea a două proteine cheie – ARN polimeraza ARN dependentă (RdRp) și helicaza. Polimerazele sunt enzime care permit sinteza lanțurilor de acizi nucleici (ADN sau ARN). Așa cum îi spune numele, RdRp permite sinteza unui lanț ARN dintr-o altă matrice ARN (prin comparație cu ARN polimeraza ADN dependentă, care produce ARN pornind de la ADN). RdRp și helicaza sunt enzime cu rol în transcrierea și replicarea coronavirusurilor.
Mecanismele de replicare și transcriere au fost studiate la coronavirusurile cunoscute. Cu toate acestea, nu era clar dacă mecanismul se aplică și la SARS-CoV-2 și dacă sunt alte elemente specifice transcriptomului.
Tehnologiile avansate de secvențiere au permis studierea transcriptomului viral
Metodele „sequencing-by-synthesis” (secvențiere prin sinteză) sunt caracterizate de o mare acuratețe. Limitarea ar fi faptul că analizează fragmente scurte, astfel încât acestea ar trebui reconstruite digital. Introducerea secvențierii directe ARN nanopore (DRS) permite citirea unor secvențe mai lungi.
Secvențierea ARN nanopore și secvențierea ADN nanoball sunt două tehnici complementare de analiză moleculare care permit studierea ARN-ului genomic și subgenomic.
Secvențierea nanopore permite analiza directă a întregului ARN viral fără a necesita fragmentarea acestuia. Secvențierea ARN convențională implică un proces alcătuit din mai mulți pași prin care molecula este fragmentată și transformată în ADN. Prin această tehnică se detectează ARN, nu ADN circular, iar analiza directă ARN se realizează în timpul secvențierii. Deși tehnica DRS are o acuratețe mai mică, permite „citirea” unor transcripte mai lungi. Poate oferi informații și despre modificările biochimice deoarece analizează ARN-ul direct.
- Prin secvențierea ARN directă, de tipul nanopore au fost identificate 41 de modificări la nivelul transcriptelor, cea mai frecventă secvență fiind AAGAA;
- Modificările epigenetice pot fi motivul evoluției rapide a virusului și ar putea oferi informații importante despre rezistența în fața atacului imun al gazdei.
Secvențierea ADN prin tehnica nanoball permite analizarea unor fragmente scurte, însă are avantajul analizării unui număr mare de secvențe cu mare acuratețe.
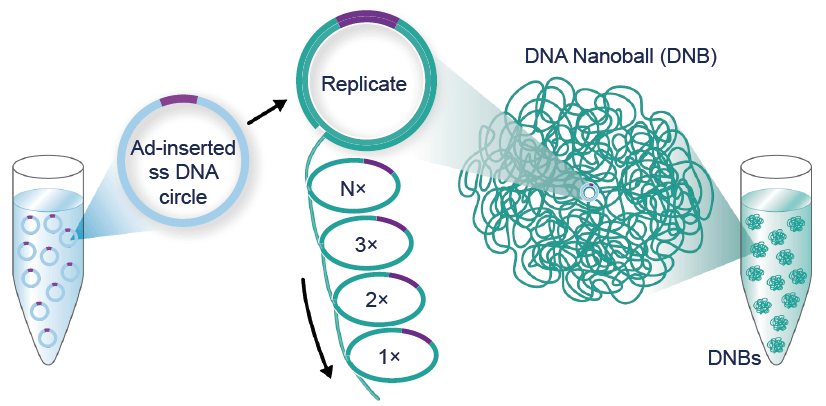
Secvențierea nanoball a oferit noi informații despre complexitatea transcriptomului:
- ARN-ul genomic și subgenomic determină transcripte care conțin ORF necunoscute până acum, apărute prin numeroase fenomene de fuziune, deleții la nivelul nucleotidelor care formează ARN-ul sau schimbarea cadrului de citire;
- Noul studiu a identificat cadre de citire exprimate doar pentru 9 molecule de ARN subgenomic și nu 10, cum se credea anterior.
Până acum au fost descrise 10 categorii de ARN subgenomic (S, E, M, N, 3a, 6, 7a, 7b, 8 și 10). În cadrul acestui studiu s-a demonstrat că ORF 10 nu este exprimat. SARS-CoV-2 exprimă 9 secvențe ARN subgenomic (S, E, M, N, 3a, 6, 7a, 7b,8).
Caracterizarea în detaliu a ARN-ului subgenomic exprimat și caracterizare ORF sunt elemente esențiale pentru a studia proteinele virale, mecanismele replicării și interacțiunile între virus și gazdă. Sinteza ARN-ului viral este complexă. Ca și ale virusuri ARN, coronavirusurile prezintă frecvent fenomene de recombinare, ceea ce permite o evoluție rapidă pentru schimbarea specificității față de gazdă, față de țesut, dar și sensibilitatea la posibile medicamente. Noile ORF identificate pot conduce la proteine accesorii care modulează replicarea virală și răspunsul imun al gazdei. Modificările ARN pot contribui la supraviețuirea virusului și la evitarea răspunsului imun în țesuturile infectate din moment ce s-a observat faptul că activarea răspunsului imun înnăscut este mai redusă față de ARN-ul care prezintă modificări nucleotidice.
Toate aceste mecanisme descoperite trebuie studiate în continuare pe modele animale și culturi celulare – dacă modificările ORF și ARN sunt unice pentru SARS-CoV-2 sau sunt conservate și la alte coronavirusuri. Studiile comparative vor aduce noi date despre mecanismele SARS-CoV-2.
Citește și
- Genomul coronavirusului SARS-CoV-2, secvențiat într-o lună de la apariția primului caz de boală. Ritmul cercetărilor este fără precedent – un vaccin ar putea fi dezvoltat în următoarele 3 luni
- #COVID-19. Analiza genomulului SARS-CoV-2: originea virusului și evoluția tulpinilor. Cum s-a transmis: liliac-pangolin-om

