#COVID-19. Analiza genomulului SARS-CoV-2: originea virusului și evoluția tulpinilor. Cum s-a transmis: liliac-pangolin-om
De la începutul anului 2020, comunitatea medicală internațională își îndreaptă atenția asupra noului coronavirus, SARS-CoV-2, și a bolii pe care o determină, COVID-19. Virusul a apărut pentru prima oară în orașul Wuhan, provincia Hubei, din China și s-a răspândit rapid la nivel global. Deoarece este un virus nou apărut, cunoașterea asupra sa și asupra COVID-19 este în dinamică, pe măsură ce noi date sunt validate și publicate. Genomul viral a fost identificat într-un timp record pentru un organism recent descoperit, în numai 30 de zile, dar studii recente sugerează că mai sunt multe detalii de înțeles despre acest virus și modul în care acționează.

Evoluția genetică a SARS-CoV-2: de unde provine, încotro se îndreaptă
SARS-CoV-2 provine dintr-o familie virală care afectează în mod majoritar animalele: printre speciile afectate se numără liliecii, porcii, șoarecii și chiar balenele beluga. Cele șapte specii de coronavirus care infectează și oamenii (dintre care cel mai recent descoperit este SARS-CoV-2) au fost identificate și la diverse specii de animale. În plus, debutul epidemiei COVID19 și primele cazuri din Wuhan au fost strâns legate de o piață agroalimentară, înainte să se răspândească prin transmitere interumană. Toate acestea sugerează că SARS-CoV-2 este un virus zoonotic, care a trecut bariera speciilor de la un animal la om. Studiile genetice au trasat drumul virusului de la gazdele animale la om, urmărind variațiile genetice ale proteinei S (spike).
Prin compararea codului genetic responsabil pentru construirea acestei proteine din diferite coronavirusuri, putem identifica originea SARS-CoV-2. Atât virusul SARS, cât și SARS-CoV-2 au afinitate ridicată pentru receptorul din celulele umane, însă structura proteinei S este semnificativ diferită între cele două virusuri. Această observație indică faptul că proteina S din SARS-CoV-2 a apărut prin selecție naturală, un proces îndelungat de acumulare de mutații genetice în generații succesive de virusuri. Ipoteza improbabilă de apariție a noului coronavirus prin manipulare umană implică utilizarea unei structuri virale deja cunoscute. SARS-CoV-2 este similar cu celelalte coronavirusuri SARS și, respectiv, MERS (care au evoluat într-o manieră epidemică în trecut) doar într-o proporție de doar 79% și, respectiv, 50%.
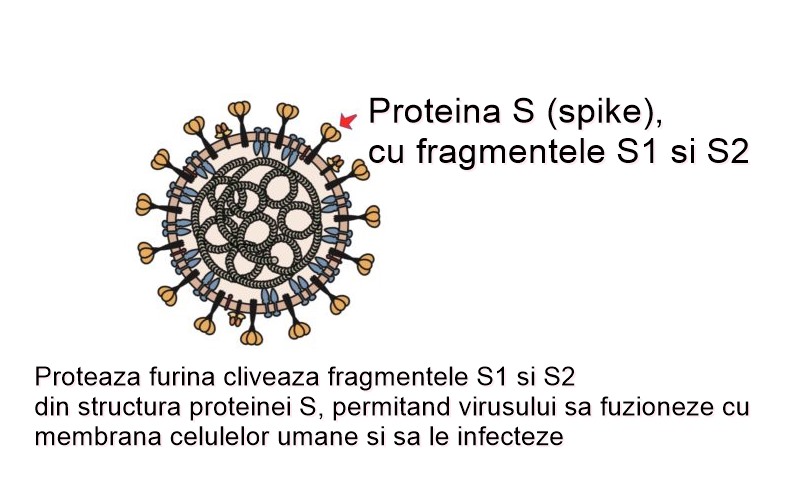
Un alt aspect care diferențiază SARS-CoV-2 de celelalte coronavirusuri cunoscute este prezența unui sit de clivare între două dintre cele trei subunități ale proteinei S. Acest fragment proteic, format dintr-o serie de aminoacizi bazici (polybasic cleavage site), poate fi clivat de furină, o enzimă care se regăsește în numeroase țesuturi din organism. La fel ca structura unică a proteinei S printre coronavirusuri, și situl de clivare este diferit de alte fragmente proteice cu funcție similară din alte coronavirusuri (virusul SARS neprezentând deloc un astfel de sit).
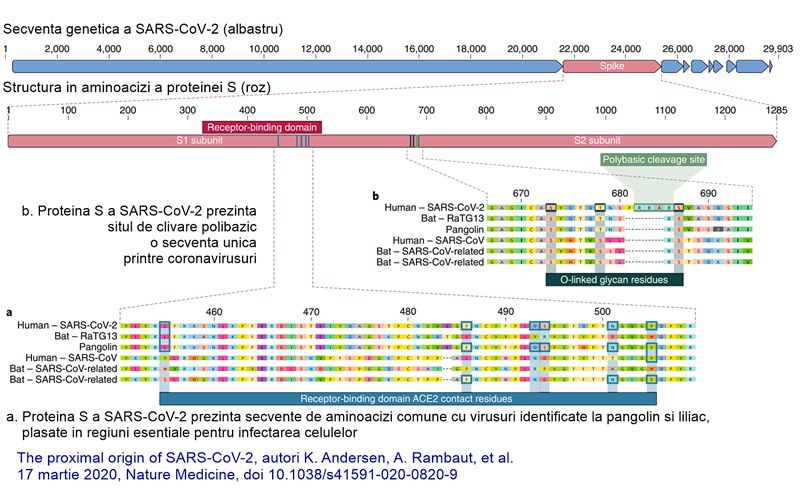
Structura genetică a SARS-CoV-2 indică liliacul drept sursa zoonotică a infecției: este identic în proporție de 96.2% cu un coronavirus din populația de lilieci din China. Totuși, fragmente importante din proteina S diferă. Gazda intermediară între liliac și om este, cel mai probabil, pangolinul, un mamifer cu corpul acoperit de solzi, care se comercializează în mod ilegal în piețele agroalimentare precum cea din Wuhan, datorită utilizării în medicina tradițională chineză. Coronavirusurile izolate de la aceste animale au în comun cu SARS-CoV-2 fragmente înalt specifice de proteină S, responsabile cu legarea de celule. Aceste două observații indică istoria SARS-CoV-2: un coronavirus specific liliecilor, care s-a transmis la pangolin în piața agroalimentară din Wuhan, acumulând mutații care cresc infectivitatea în acest proces.
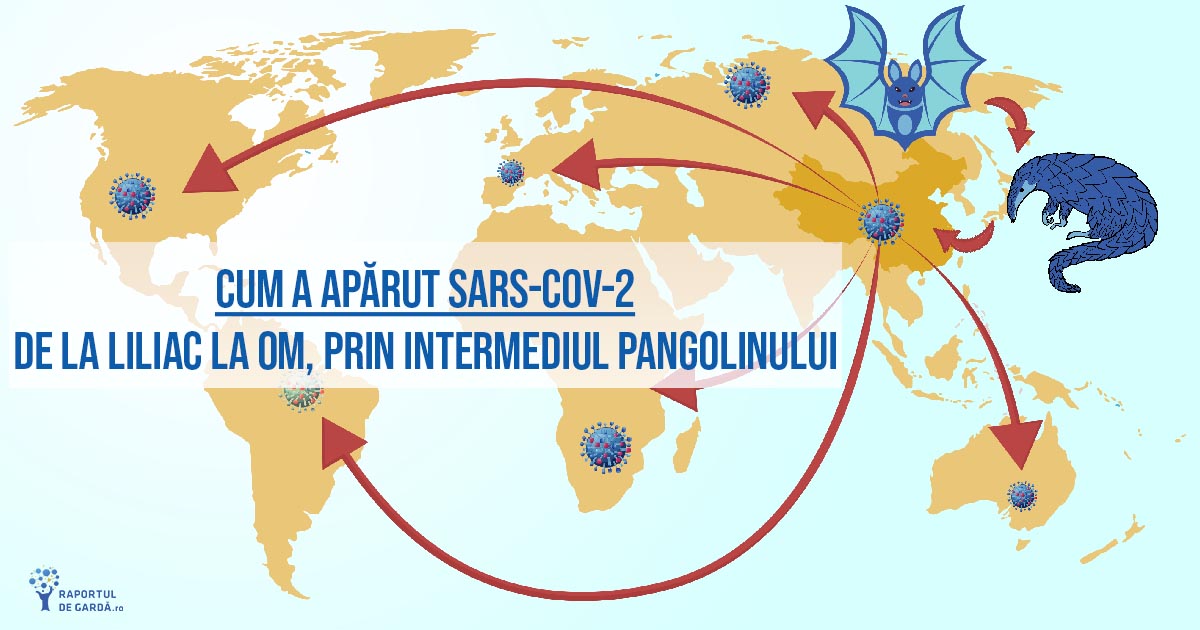
Mai recent, cercetările au confirmat originea zoonotică prin identificarea „verigii-lipsă”: specia de coronavirus care infectează pangolinul. Prin analiza unor fragmente tisulare, s-a identificat genomul unui coronavirus întâlnit în mai multe probe, și a fost comparat cu coronavirusul specific liliecilor și cu SARS-CoV-2. Diferite secvențe de nucleotide au fost identice cu SARS-CoV-2 în proporții variind între 85,5% și 92,4%. Cel mai semnificativ aspect este însă similaritatea înaltă, de 97,4%, în fragmentul genetic corespunzător proteinei S. Fragmentul responsabil cu interacțiunea cu receptorul uman (ACE2) este identic într-o proporție de 84,8% între virusul din pangolin și SARS-CoV-2, față de 80,8% – 81.4% între virusul din liliac și SARS-CoV-2, indicând o legătură filogenetică mai apropiată.
Astfel, originea zoonotică este mult mai probabilă decât cea ”de laborator”: în primul rând, în laboratoarele care cercetează coronavirusuri nu s-au raportat prezența de specii suficient de similare cu SARS-CoV-2 încât să explice apariția acestuia, iar fragmentul comun al proteinei S între SARS-CoV-2 și coronavirusul specific pangolinului indică această cale de apariție drept mult mai probabilă.
Comportamentul SARS-CoV-2 de la începutul epidemiei COVID19
Compararea proteinei S cu proteina S a altor coronavirusuri relevă o altă observație, cu semnificație epidemiologică: capacitatea SARS-CoV-2 de a se lega de celulele umane este superioară altor coronavirusuri.
În primul rând, existența sitului polibazic de clivare (menționat anterior), la nivelul căruia poate activa o enzimă ubicuitară din organism (furina) îi conferă noului coronavirus capacitatea de a infecta, în mod eficient, mai multe țesuturi diferite. Prin clivare, fragmentele din proteina S responsabile cu pătrunderea în celulă sunt activate. Enzima furină este, în plus, bogat distribuită în tractul respirator și în țesutul pulmonar.
SARS-CoV-2 și SARS se atașează de același receptor în momentul infecției gazdei umane – enzima de conversie a angiotensinei (ACE2). Totuși, comparația genetică relevă faptul că proteina S de suprafață (care interacționează cu ACE2) a celor două virusuri diferă în mod semnificativ, iar proteina S a SARS-CoV-2 are o afinitate superioară față de receptorul uman. Aceste diferențe se datorează, probabil, recombinării genetice suferite de virus în cadrul gazdei sale intermediare, pangolinul.
Un aspect important, care poate indica schimbări în comportamentul virusului în organismul uman, este prezența de mutații genetice. Pe măsură ce virusul infectează mai mulți oameni și se multiplică, există posibilitatea apariției de mutații, care să conducă la tulpini virale distincte, cu comportamente diferite, în ceea ce privește infectarea de noi pacienți, transmiterea interumană, agresivitatea formelor clinice de COVID19. Până în acest moment, cercetările indică un grad scăzut de variabilitate genetică în cazul SARS-CoV-2.
Astfel, este necesară analiza comparativă a genomului viral prelevat de la pacienți din regiuni și momente diferite ale epidemiei, pentru a identifica diferențele genetice. Prin compararea a 103 probe, s-a observat că există două tulpini distincte de coronavirus SARS-CoV-2, denumite L și S (în funcție de aminoacidul codificat de regiunile omoloage diferite ale celor două tulpini). Se estimează că 70% dintre pacienții COVID19 sunt infectați cu tulpina L, iar 30% cu S. Reconstruind evoluția celor două tulpini, se observă că S se regăsește de mai multă vreme în circulație, iar tulpina L a apărut ulterior, evoluând din tulpina S.
Variabilitatea genetică, explicație pentru agresivitatea virusului?
În primul rând, este important de amintit că SARS-CoV-2 nu conduce la o proporție ridicată de cazuri severe de pneumonie, și nici la o mortalitate crescută, în comparație cu gripa sezonieră. Mai exact, 80% dintre cazuri sunt ușoare, 15% sunt severe și doar 5% sunt grave, necesitând suport ventilator. În ceea ce privește mortalitatea, până în acest moment, 3.4% dintre cazurile raportate OMS au evoluat către deces. Această valoare este mai mare decât în cazul gripei sezoniere, care are o mortalitate de maximum 0,15% (conform estimărilor CDC pentru iarna 2019-2020), însă net inferioară mortalității cauzate de coronavirusurile care au condus la epidemii în trecut, SARS și, respectiv, MERS, cu mortalități de 9.6% și, respectiv, 34%.
Având în vedere faptul că tulpina L este predominantă (70%), deși a apărut mai recent, și, în plus, prezintă o variabilitate genetică mai mare, putem deduce că se transmite și se replică mai rapid în populația umană. Acest comportament îi permite să acumuleze mutații la fiecare replicare, și să se răspândească mai rapid.
Primii pacienți diagnosticați, din Wuhan, au fost infectați preponderent cu tulpina L (96.3%). Ulterior, pacienții din restul Chinei, au prezentat tulpina L într-o proporție de doar 61.6%. Acest lucru poate fi explicat de măsurile prompte ale autorităților: prin izolarea și carantinarea precoce, transmiterea tulpinii L, mai rapidă și agresivă, a fost stopată, iar tulpina S, mai lentă, a putut atinge o incidență mai mare.
Toate aceste concluzii reprezintă, însă, rezultatele analizei unei proporții mici de probe din totalul infecțiilor SARS-CoV-2. O analiză similară este necesară și în alte regiuni afectate de COVID19, precum Europa sau SUA. Cercetări asupra probelor virale de la pacienți din Italia confirmă faptul virusul SARS-CoV-2 nu are o tendință pronunțată spre a acumula mutații genetice: în populația italiană au fost descrise doar cinci variante genetice virale, un număr scăzut pentru un număr atât de mare de cicluri replicative.
Pentru informațiile corecte și verificate, accesați și stirioficiale.ro, primul website lansat de Code for Romania Task Force, în parteneriat cu Guvernul României prin Autoritatea pentru Digitalizarea României și Departamentul pentru Situații de Urgență.
Citește și:
- Pacienții cu boli cardiovasculare și diabet au risc crescut de evoluție severă a infecției cu noul coronavirus SARS-CoV-2
- EFPIA și-a anunțat angajamentul în lupta împotriva COVID-19. Ce soluții caută companiile farmaceutice europene împotriva noului coronavirus
- #SurseOficiale: Ghid pentru cumpărături în timpul pandemiei COVID-19. Ce trebuie să facem înainte, în timpul și după efectuarea cumpărăturilor?

